Rapp. Comm. int. Mer Médit., 37,2004
274
SPATIAL AND TEMPORAL SCALES FOR THE MONITORING
OF BACTERIAL COMMUNITY STRUCTURE IN COASTAL ECOSYSTEMS.
J.F. Ghiglione*, M. Larcher, S. Emonet, P. Lebaron
Observatoire Océanographique de Banyuls - UMR CNRS 7621
Université Paris VI. Banyuls Sur Mer, France - * ghiglion@obs-banyuls.fr
Summary
One of the challenges in microbial ecology is to determine at what temporal and spatial scales the structure of natural communities should
be monitored. Samples of different volumes and taken at different spatial (vertical and horizontal) and temporal (from hours to seasons)
scales were analyzed to investigate at which scale one should monitor community structure at a coastal station. Investigations were
performed in the Bay of Banyuls (Mediterranean, France). No changes in the community structure was detectable within a radius of 200
meters around the station whereas changes were found at both weekly and monthly scales.
Keywords : marine bacterial communities, diversity, sampling strategy
The last decade of microbial ecology has resulted in a veritable
explosion of studies that use new molecular biological techniques to
analyse the structure of microbial communities. These studies have
improved our perception of microbial diversity and community
composition in marine ecosystems (1). However, the temporal and
spatial scales at which bacterial populations vary remained poorly
documented. A comparison of recent studies of bacterial community
dynamics in pelagic marine waters showed minimal differences in
community compositions at stations a few miles apart near Anvers
Island, Antartica (2) or 1500 km apart in the Arabian Sea (3), in
opposition to clear differences observed along the Catalan Coast,
Spain (4). Short-term studies showed major changes in bacterial
communities consequent to qualitative changes in the pool of organic
matter (5, 6). These evidences for space- and time-dependent bacterial
community changes emphasized the importance of sampling stra-
tegies when studying the dynamic of natural communities.
Although a large set of methodologies have been developed to
investigate the structure of bacterial communities, the selection of the
most appropriate method depends on the questions to be answered and
on the amount of samples to be processed within a reasonable period
of time. Fingerprinting approaches offer the best compromise for the
monitoring and comparison of microbial assemblages and for the
assessment of temporal and spatial changes that would not have been
feasible using time-intensive 16S rDNA sequence analysis.
Denaturing gradient gel electrophoresis (DGGE) has become a very
popular fingerprinting technique in marine microbial ecology,
especially because bands with particular melting behaviour can be
excised from the gel and subsequently sequenced to reveal the
phylogenetic affiliation of the community members (7). However, its
laborious technical optimisation including calibration of the linear
gradient of DNA denaturants that makes gel-to-gel comparison
difficult have made this technique impractical for the fine comparison
of more than samples loaded on one gel (8). The recently developed
capillary electrophoresis-single strand conformation polymorphism
(CE-SSCP) fingerprinting technique permitted high reproducibility
for reliable comparison of patterns from a theoretically infinite num-
ber of samples. Because a size standard with a different ?uorescent
label is added to each sample, CE-SSCP and further computing
correction encompassed the problem of gel-to-gel comparison (9).
In this study, CE-SSCP technique was used to determine spatial and
temporal scale changes in the structure of bacterial communities in a
coastal ecosystem. Samples of different volumes and taken at
different spatial (vertical and horizontal) and temporal (from hours to
seasons) scales were analyzed to investigate at which scale one should
monitor community structure at a coastal station. Investigations were
performed in the Bay of Banyuls (Mediterranean, France).
No changes in the community structure was detectable within a
radius of 200 meters around the SOLA station but changes occurred
at larger scales. Changes were found at both weekly and monthly
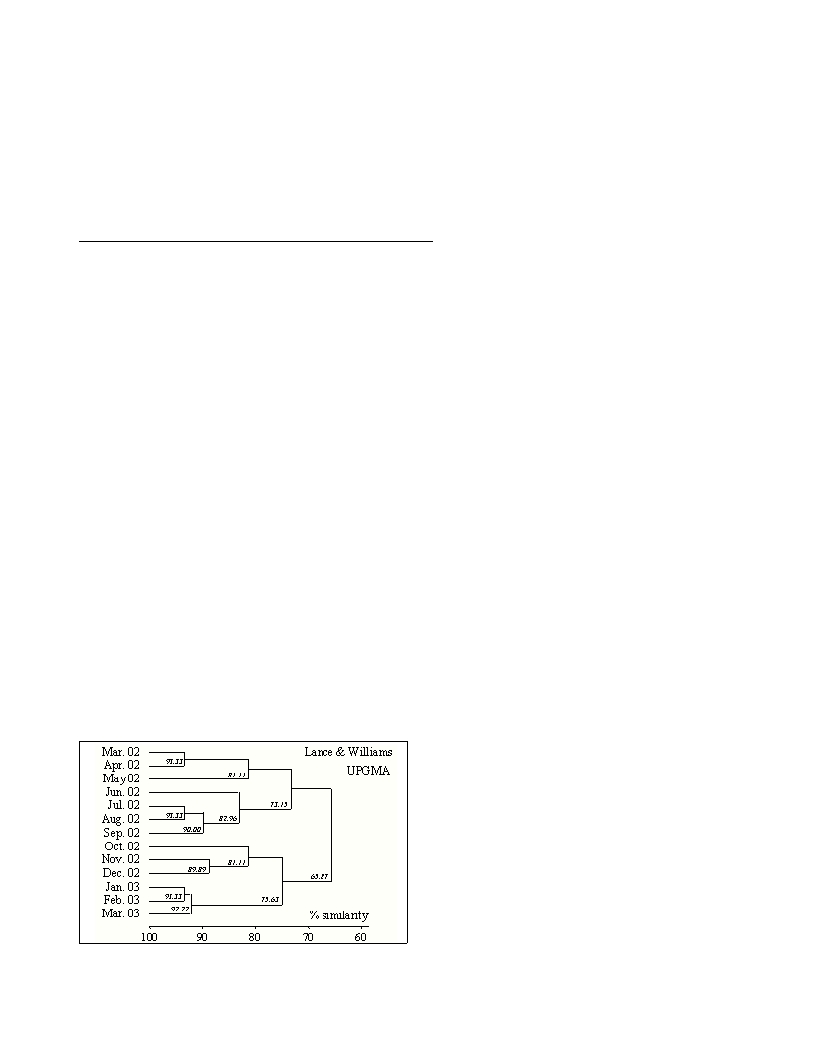
scales. Cluster analysis based on a one-year monitoring at the SOLA
station showed changes in the seasonal distribution of
bacterioplankton community structure, without any return to the
initial community structure after one year (Fig. 1).
References
1-Giovannoni S., Rappé M., 2000. Evolution, diversity, and molecular
ecology of marine prokaryotes. Pp. 47-84. In: Kirchman DL (ed.)
Microbial ecology of the oceans. Wiley-Liss, New York.
2-Murray AE, Preston CM, Massana R, Taylor LT, Blakis A, Wu K,
DeLong EF, 1998. Seasonal and spatial variability of bacterial and
archaeal assemblages in the coastal waters near Anvers Island, Antarctica.
Appl. Environ. Microbiol.,64: 2585–2595.
3-Riemann L, Steward GF, Fandino LB, Campbell L, Landry MR, Azam
F, 1999. Bacterial community composition during two consecutive NE
Monsoon periods in the Arabian Sea studied by denaturing gradient gel
electrophoresis (DGGE) of rRNA genes. Deep-Sea Res. II, 46:
1791–1811.
4-Schauer M, Massana R, Pedrós-Alió C, 2000. Spatial differences in
bacterioplankton composition along the Catalan coast (NW
Mediterranean) assessed by molecular fingerprinting. FEMS Microbiol.
Ecol.,33:51–59.
5-Fandino L.B., Riemann L., Steward G.F., Long R.A., Azam F., 2001.
Variations in bacterial community structure during a dino?agellate bloom
analyzed by DGGE and 16S rDNA sequencing. Aquat. Microb. Ecol.,23:
119–130.
6-Schäfer H., Bernard L., Courties C., Lebaron P., Servais P., Pukall R.,
Stackebrandt E., Troussellier M., Guindulain T., Vives-Rego J. and
Muyzer G., 2001. Microbial community dynamics in Mediterranean
nutrient-enriched seawater mesocosms: changes in the genetic diversity of
bacterial populations. FEMS Microbiol. Ecol.,34: 243-253.
7-Muyzer, G., Brinkhoff, T., Nübel, U., Santegoeds, C., Schäfer, H. and
Wawer, C., 1998. Denaturing gradient gel electrophoresis (DGGE) in
microbial ecology. Pp. 1-27. InA.D.L. Akkermans, J.D. van Elsas, and F.J.
de Bruijn (ed.), Molecular microbial ecology manual, vol. 3.4.4. Kluwer
Academic Publishers, Dordrecht, The Netherlands.
8-Moeseneder M.M., Arrieta J.M., Muyzer G., Winter C., Herndl G.J.,
1999. Optimization of terminal-restriction fragment length polymorphism
analysis for complex marine bacterioplankton communities and
comparison with denaturing gradient gel electrophoresis. Appl. Environ.
Microbiol.,Aug. 65(8): 3518-25.
9-Zumstein E., Moletta R., Godon J.J., 2000. Examination of two years
of community dynamics in an anaerobic bioreactor using ?uorescence
polymerase chain reaction (PCR) single-strand conformation
polymorphism analysis. Environ. Microbiol., 2:69-78.
Fig. 1. Cluster analysis dendrogram based on comparison of 16S PCR-
CE-SSCP patterns from one year sampling at the SOLA station
(Mediterranean, France). The tree was constructed by applying
unweighted pair group average linkage rules on a Lance and Williams
index of similarity matrix calculated from the CE-SSCP profiles.