Rapp. Comm. int. Mer MÚdit., 36,2001
364
Most animal species often show a subdivision into local populations (1).
If local populations are exposed to contrasting environmental conditions
appreciable populations differentiation may arise determined by genotype-
environment interactions (1). Theoretical works provide that populations
from unpredictable environment, present a general reduction of genetic
variability (2, 3). In order to provide insights on the relationship between
genetic structure and habitat type of animal populations we investigated
the degree of allozyme genetic dive
rgence between individuals of
Mytilaster minimusPoli, 1795 (Mollusca, Pelecypoda). M. minimusis a
small bibalve with maximum shell length of roughly 2 cm, distributed on
hard bottoms in lagoons and marine sites of the intertidal belt in the
Mediterranean and eastern Atlantic. Life cycle has a planktonic larval stage
but larval morphology and life-span are unknown, so that its potential for
dispersal cannot reliably be inferred.
Eleven samples (about 40 specimens each) were collected in four dif-
ferent brackish-water basins in western Sardinia (Italy) and in the nearest
rocky shore. Whenever possible, more samples were collected within each
basin from the inner part of the ponds toward the sea. Five of eleven sam-
ples have been collected in the ponds, 3 in the sea and 3 in intermediate
sites. Allozyme cellulose acetate electrophoresis, carried out using the pro-
cedures outlined in a previous study (4), gave the allele frequencies at 15
presumptive loci. The genetic variability was estimated by the average
expected heterozigosity over loci (H). The genetic structure of the whole
set of populations was analysed by F-statistics. Unbiased estimators were
obtained by jackknifing across loci and tested for difference from zero by
permutation tests. The level of genetic distance between populations was
estimated with Nei's (5) unbiased index, D.Multidimensional scaling
(MDS) was applied to the matrix of genetic distances.
Our results showed a general devi-
ation from Hardy-Weinberg expecta-
tions at all loci over all populations
(F
IS
=0.532, P< 0.001). Approxima-
tely, the observed heterozigosity is
one half of the expected one.
Deficiency of heterozygote individu-
als has been commonly observed in
allozyme studies on marine and
brackish-water bivalves and re?ect a
trend characteristic to invertebrates
with high rate of reproduction and
high dispersal capability (6, 7. 8). In
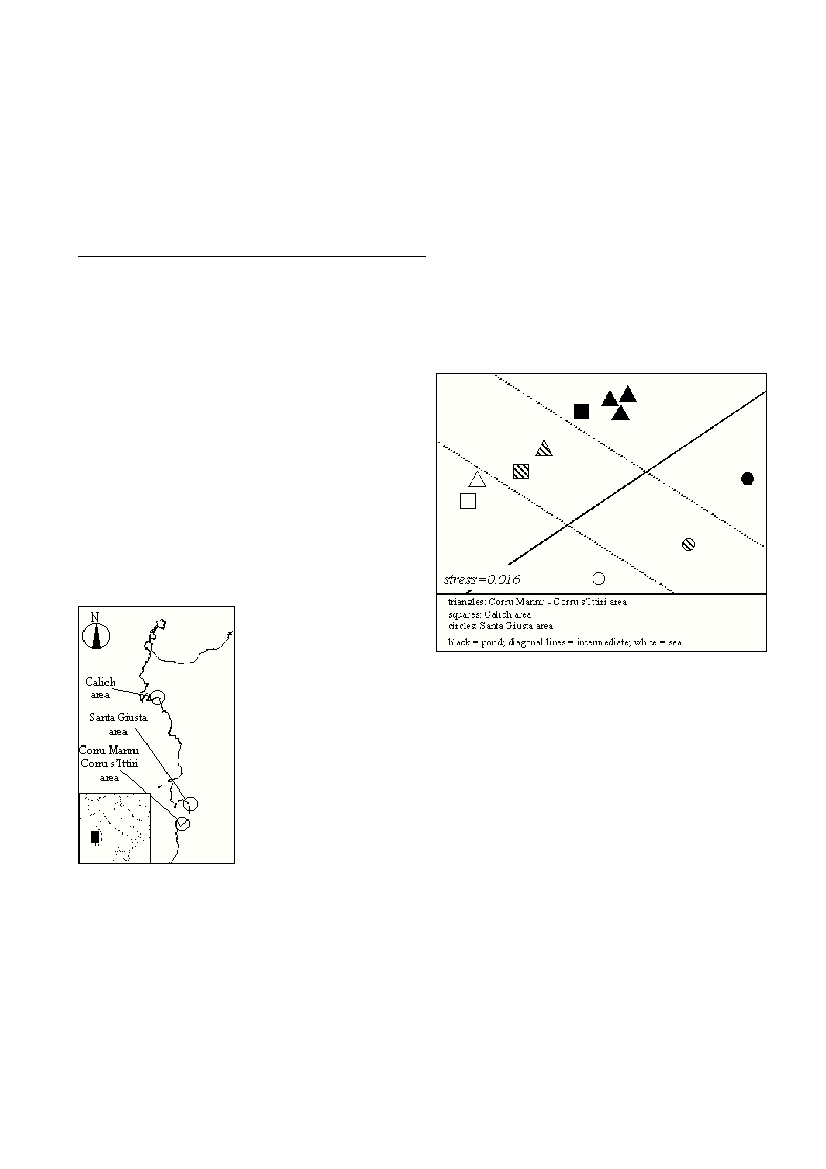
the MDS plot the straight line clearly
separates the Santa Giusta samples
from the others (Fig. 2) while dotted
lines separate three groups of sam-
ples that are consistent with the gra-
dient pond-sea (Fig. 2). The diver-
gence of Santa Giusta samples from
the others could be a consequence of
ecological peculiarity of that site
and/or depend on the effect of genet-
ic drift after recurrent summer dys-
trophic crises. The genetic distances
found between M. minimus samples
are below the values detected for
congeneric species (for a review see 9). The highly significant value of the
coancestry coefficient (F
ST
=0.239, P<0.001) referred to the eleven popula-
tions remark their high degree of genetic divergence. The occurrence of
genotypes typical of each habitat-type accounts for the observed genetic
divergence. Furthermore a number of habitat-private alleles were detected.
In addition, in some instances the most frequent allele present in brackish-
water samples is the less common in marine samples and vice versa. Our
results can be explained in terms of adaptation and local selection. The
selective forces acting in each habitat-type favour the different genotype
and the genetic divergence observed. On the other hand, geographical dis-
tance and/or history of colonisation contributed to the present patterns of
genotypic variability.Thus the observed pattern results from the superim-
position of two main types of forces that in?uenced population differenti-
ation in a process of local adaptation since the first colonisation events of
brackish-water habitats.
Figure 2. . Multidimensional scaling of Nei's genetic distances. The
low value of stress indicates that distances among samples on the plot
represent the genetic distances accurately.
References:
1 - Carvalho G.R., 1993. Evolutionary aspects of fish distribution: genetic
variability and adaptation. J. Fish Biol., 43(suppl. A):53-73.
2 - Battaglia B. and Bisol P.M., 1988. Environmental factors, genetic
differentiation, and adaptive strategies in marine animals. pp.393-410. In:
Rotchshild B.J. (ed.), Toward a theory on biological-physical interactions in
the world ocean. Kluwer Acad. Publ., The Netherlands,
3 - Cognetti G. and Maltagliati F., 2000. Biodiversity and adaptive
mechanisms in brackish water fauna. Mar. Pollut. Bull., 40:7-14.
4 - Camilli L., 2000. DiversitÓ genetica di popolazioni di invertebrati di
biotopi marini e salmastri. Ph.D. Thesis, University of Pisa, Italy, pp.1-121.
5 - Nei M., 1978. Estimation of average heterozygosity and genetic distance
from a small number of individuals. Genetics, 89:583-590.
6 - Johnson M.S. and Black R., 1984. The Wahlund effect and the
geographical scale in the intertidal limpet Siphonaria. Mar. Biol., 79:295-302.
7 - Volckaert F. and Zouros E., 1989. Allozyme and physiological variation in
the scallop Placopecten magellanicusand a general model for the effects of
heterozygosity on fitness in marine molluscs. Mar. Biol., 103:51-61.
8 - Borsa P., Zainuri M. and Delay B., 1991. Heterozygote deficiency and
population structure in the bivalve Ruditapes decussatus. Heredity,66:1-8.
9 - Thorpe J.P. and SolÚ-CavaA.M., 1994. The use of allozyme
electrophoresis in invertebrate systematics. Zool. Scri., 23:3-18.
ALLOZYMIC GENETIC DIVERGENCE IN THE BIVALVE MYTILASTER MINIMUSFROM
BRACKISH-WATER AND MARINE HABITATS IN THE WESTERN SARDINIAN COAST (ITALY)
Laura Camilli
1
*, Alberto Castelli
2
, Claudio Lardicci
1
, Ferruccio Maltagliati
1,2
1
Dipartimento di Scienze dell'Uomo e dell'Ambiente, Pisa, Italy - camilli@discau.unipi.it
2
Dipartimento di Zoologia e Antropologia, Sassari, Italy
Abstract
The genetic relationships among 11 samples of the bivalveMytilaster minimusfrom brackish-water and marine habitats along western
Sardinian coast were studied by allozyme electrophoresis. Results obtained for 15 loci showed high levels of both within- and between-
population genetic heterogeneity.The positive and significant value of the inbreeding coefficient indicated a general departure from HW
equilibrium due to deficiency of heterozygotes. The observed patterns of genetic variability result from the superimposition of two main
evolutionary forces: 1) the diverse selective regimes in the different habitat-types and 2) the different history of colonisation and/or
geographical distribution.
Keywords: bivalves, brackish water, genetics, Western Mediterranean
Figure 1. Mytilaster minimus.
Location of sampling areas.
Within each area samples from
b
r
a
c
k
i
s h -
w
a t
e r, marine and